Przydatność DNA mitochondrialnego w genealogii
Burzliwy rozwój Genealogii
Genetycznej w ciągu ostatnich 10 lat przyniósł wiele zaskakujących informacji i
dał narzędzia do rozmaitych analiz genealogicznych. Genealogia Genetyczna – aczkolwiek
może być używana również w medycynie, jest z gruntu dyscypliną pomocniczą
Historii, w szczególności – Genealogii.
W ostatnich latach
udostępnione zostały – przez zmianę technik sekwencjonowania DNA – analizy Pełnego
Genomu mitochondrialnego DNA (FGS mtDNA). Te DNA reprezentuje linie
żeńską/matczyną przenoszone jest bowiem na dzieci wyłącznie przez matkę. DNA
mitochondrialne (mitochondrium to autonomiczna organella wewnątrzkomórkowa odpowiedzialna
za procesy oddychania na poziomie komórki). Ponieważ odpowiada za oddychanie,
wytwarzanie energii – więc jest dość mało podatne na mutacje, a te które
powstają często nie mogą być utrwalone. Poza tym mtDNA zawiera jedynie
16 tysięcy par nukleotydów (16 kbp) podczas nawet mały chromosom Y warunkujący
płeć męską i charakterystyczny dla linii ojczystej zawiera już 30 milionów par
nukleotydów (30 Mbp= 30.000 kbp). Tym niemniej, ponieważ kod genetyczny DNA
jest zdegenerowany tzn. za kodowanie poszczególnych aminokwasów tworzących
białka – tu: enzymy mitochondrialne - odpowiadają czasem 2-3 kombinacje nukleotydów więc jest pole do
pewnej ilości mutacji obojętnych biologicznie. I właśnie gromadzeniem się
takich mutacji zajmuje się Genealogia Genetyczna.
Mutacje powstają w zasadzie w równym i nieśpiesznym
tempie. Proces różnicowania linii w przypadku DNA mitochondrialnego to tysiące
lat, kiedy Y-DNA zmienia się w tempie setek lat. Badając różnice między
poszczególnymi liniami macierzystymi spostrzeżono, że zbiegają się one do
wspólnej przodkini a z kolei poszczególne linie też posiadają wspólnych
przodków, aż do jednej pramatki wszystkich linii macierzystych. To tzw.
mitochondrialna Ewa. Według współczesnych wyliczeń żyła w Afryce między 140-200
tysięcy lat temu, a więc już po wyodrębnieniu się anatomicznie współczesnego gatunku
Homo Sapiens 190-200 tysięcy lat temu. Jej sekwencja mtDNA opisana jest skrótem
RSRS, do tego standardu porównujemy wszystkie mtDNA.

Co ciekawe, na
mitochondrialnej Ewie nie kończy się analiza mtDNA, możemy porównywać DNA
do innych gatunków ludzkich, wymarłych ok. 40 tys. lat temu – neandertalczyka i
denisowiańczyka. Zbiegają się one z kolei we wspólnej przodkini rodzaju Homo,
ok. 900 tysięcy lat temu, kiedy człowiek nauczył się korzystać z ognia.
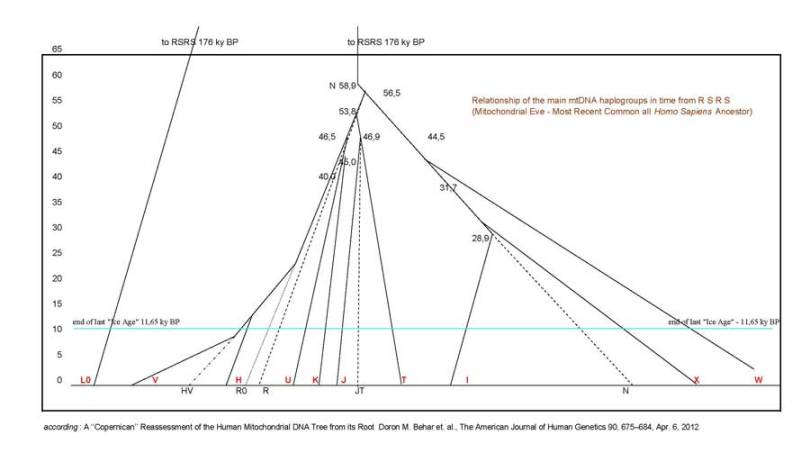
Oczywiście podziała na
wiele linii rozwojowych współczesnego człowieka miał miejsce już po wyjściu z
Afryki i zasiedleniu Eurazji ponad 50 tysiecy lat temu. Widoczna na schemacie
pozioma linia przedstawia koniec ostatniej Ery Lodowcowej, kiedy ukształtowane
już były wszystkie gałęzie opisane literami, w tym pochodzące od „7 córek
Europy” o imionach Urszula, Xenia, Helena, Velda, Tara, Katarzyna, Jasmina.
Oczywiście, jeśli dwie
osoby należą do różnych gałęzi czyli haplogrup to nie mogą być spokrewnione w
linii żeńskiej. Takie ustalenie od razu weryfikuje negatywnie niektóre
hipotetyczne powiązania genealogiczne. Pozytywna weryfikacja jest trudniejsza,
zwłaszcza w przypadku popularniejszych haplogrup, np. H. Ale jeśli u kogoś
spodziewamy się rzadkiej haplogrupy X i okazuje się ją mieć – to jest bardzo
duża szansa na potwierdzenie pokrewieństwa (X to zaledwie 2-3% populacji
polskiej lub niemieckiej).
Jeśli nie opierać się tylko
na wstępnej analizie części sekwencji mtDNA a zainwestować w oznaczenie pełnej
sekwencji mtDNA to ukażą się nam szczegółowe podgałęzie macierzystego drzewa
rodowego sięgające ostatniego tysiąclecia, zob. http://www.mtdnacommunity.org/human-mtdna-phylogeny.aspx
Co więcej, w kierowanych przeze mnie projektach, np. Baltic Sea DNA Project (⅔
tysiąca uczestników z całego świata) zdarzają się osoby, które mają identyczne
regiony szybkomutujące mtDNA widoczne w tabelach projektów, a różniące się
mutacjami w tzw. Kodującym Regionie (CR), w którym zapisywane są białka enzymów
odpowiedzialnych za procesy życiowe – więc nie ujawniane publicznie. I tak np.
osoby należące do podgałęzi H2a1c powstałej
ok. 1.800 lat temu (z szerokim
marginesem błędu) na pewno są krewnymi w okresie od powstania tej gałęzi do
teraz. A analiza FGS mtDNA pozwala na ocenę pokrewieństwa w czasach
historycznych. Jednak
#97423 Lassek ma 0 mutacji w CR
#286537 Butwicka ma 1 mutację w
CR a
#183916 Krzyżanowski ma 2 mutacje w CR,
obie inne od występującej u Butwickiej.
Zatem wszystkie 3 osoby
mają wspólną przodkinię owe 1800 lat temu, kiedy powstała mutacja G3834A położona
w regionie kodującym. Z tym, że pan Lassek więcej nie wyciągnie ze swojego
mtDNA. Może stwierdzić jedynie 100% zgodność mtDNA i oznaczać to będzie, że
osoby z nim zgodne miały wspólną przodkinię w okresie od II do XX wieku.
Natomiast fakt, że z panem Krzyżanowskim pokazały się w bazie FTDNA całkowicie
zbieżne genetycznie osoby (mające wspólne mutacje prywatne) wskazuje na
pokrewieństwo w czasach kiedy, istniały już źródła historyczne, zapewne mniej
niż 600 lat temu.
Ciekawą analizę
szczegółowych pokrewieństw w mojej gałęzi X2e1 przeprowadziłem przez
zaproszenie do testów kuzynów z różnych linii. Okazało się, że istnieje 100% zgodność mutacji u wszystkich
potomków mojej praprababci Maurycji Śniechowskiej (1839-1914). Jej linia
macierzysta, a zatem i nasza, prowadzi do jej prapraprababki Konstancji z
Lipskich (1696-1758), której rodzony brat, kardynał Jan Aleksander Lipski,
książę arcybiskup krakowski, pochowany został w kaplicy Lipskich w Katedrze
Wawelskiej.
Zatem dochodzimy do tu do ważnej
cechy ustaleń genealogicznych – to obiektywne, spoza klasycznej genealogii
papierowej, potwierdzenie jej ustaleń. Tak potwierdzono identyfikację szczątków
zabitego w 1485 r. króla Anglii Ryszarda III, pochowanego w zlikwidowanym 100
lat później kościele Franciszkanów w Leicester.
Po zidentyfikowaniu
prywatnych mutacji mtDNA w naszej rodzinie (wszystkie są jak napisałem
utrwalone i bez znaczenia biologicznego) okazało się, że znalazła się osoba –
p. Szulecki #200042, którego linia macierzysta sięga połowy XVIII wieku i
kieruje się w kierunku Wielkopolski, jest odrębna od naszej, a jednocześnie występuje
u niego brak 2 szybkozmiennych mutacji, pomijanych przy tworzeniu drzewa
filogenetycznego dodatkowych mutacji:
Kolejną sensacją okazał się
wynik analizy FGS pani Kennedy z USA, o korzeniach bawarskich, z okresu
porównywalnego do naszej najdalej zidentyfikowanej przodkini w linii żeńskiej,
Zofii Szczypierskiej spod Kalisza, ur. ok. 1580 r. Był identyczny prócz tego że
zawierał tylko jedną dodatkową mutację 309.1C. Tym wynikiem osiągnęliśmy
właściwie kres możliwości metody i identyfikując przy okazji środkowo -
europejski subklad charakteryzujący się mutacją
G6962A.
Podsumowując, metoda
genealogii mtDNA jest bardzo wartościowa do potwierdzania pokrewieństw w
liniach żeńskich, w niektórych przypadkach nawet do identyfikacji krewnych. W
przypadku osób które nie wiedzą jaką charakterystykę genetyczną ma ich mtDNA
gorąco polecam decydowanie się od razu na kompletne oznaczenie za ok. 150$ bo
tylko ono daje pełną wiedzę i podstawę do interpretacji a nawet znalezienia
kuzynów.
Stanisław
J. Plewako
powyższy tekst opublikowany został w półroczniku "Dziedzictwo Kresowe", nr 7 (grudzień 2015 r.) ISSN 2299-7024